| 本文中,我们会为老师详细解析在做完m6A测序后,如何对差异甲基化的基因进行验证。简单来说就是IP后如何进行qPCR 实验。文中的表述:MeRIP-qPCR与m6A-IP-qPCR等同。
【确定样本起始量和IgG】
在进行讲解之前,我们要回答的第一个问题就是,用多少RNA来做后期验证呢?举个例子,假如要验证10个基因,究竟需要多少total RNA或polyA的RNA才合适呢?
我们先进行一个简单的计算,常规的RT-qPCR 1个反应(1个复孔)需要的total RNA至少0.1-1μg左右。我们按照最小反应起始量来算的话,那么1个反应需要至少100ng的total RNA,3个技术重复(3个复孔),总计total RNA为300ng。
我们知道total RNA中绝大部分为rRNA,那么只有不到5%的RNA为我们需要研究的对象。PCR反应需要引物跟目标序列区域进行充分碰撞,那么如果只使用mRNA会大大降低一次PCR反应的起始量。通过计算我们认为5-10ng做1次PCR反应较为合理。也就是1个样本中验证1个基因不做生物学重复只做3个复孔需要至少15ng的mRNA或300ng的total RNA。那么3个生物学重复就需要9个反应,总计45-90ng mRNA或900ng的total RNA。
根据上面的推算,IP下来的mRNA若有500ng,可以验证6-12个基因左右(1个基因3个生物学重复,每个重复3个复孔即技术重复,所以一个基因总计9次反应)。哺乳动物IP效率在10-20%左右,植物在20-40%左右(最高可达50%)。那么假设要验证6-10个基因,哺乳动物需要至少2.5-5μg的mRNA和100-200μg的total RNA,植物样本至少需要1.25-2.5μg的mRNA和50-100μg的total RNA。若加入IgG抗体这个步骤,则需要对total RNA的起始量再次翻倍。
但是如果total RNA太少该怎么处理呢?没关系,可以直接用m6A抗体对total RNA进行IP,而且不用打断。
那么接下来在进行讲解前的第二个问题就是,明明我们做高通量测序只有一个input文库和一个IP文库,为何我们做MeRIP-qPCR 要加入IgG抗体呢(如下图所示)?;
那是因为高通量测序可以对所有被m6A抗体富集下来的RNA进行全转录层面的扫描分析,一旦m6A抗体有非特异性富集,在数据呈现上我们可以明显发现reads的分布比较乱且没有规律,而特异性富集则reads几乎富集在哺乳动物的3’ UTR区和植物的5’ UTR和3’ UTR区。所以这就是测序不需要IgG文库,而我们做m6A-IP-qPCR 需要IgG富集的原因。
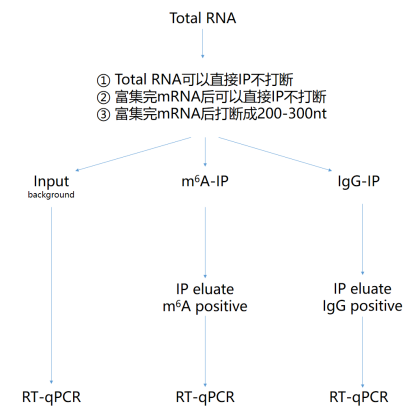
搞定了样本起始量和IgG的问题后,我们具体来看下步骤:
【步骤详解】
第一步,先对RNA进行特异性富集。至于打断或不打断,都是可行的。假如要打断的话请在打断之前,确认研究对象只是mRNA还是lncRNA。若只研究mRNA 可以先用oligodT磁珠进行富集后再进行打断。如果是lncRNA和mRNA都要研究,则需要先用试剂盒将total RNA的rRNA先消化干净,再进行打断。如果是进行高通量测序打断长度约为100nt左右,而qPCR 则需要长度至少在200nt以上,部分论文甚至打断长度在300-500nt左右甚至都不打断。一般根据文献中的资料显示,IP下来的片段用引物扩增出来的长度在130-150左右,所以100nt不到的长度要设计出合适的引物会非常困难。除非是用5’RACE扩增,否则还是建议大于200nt。当然了,如果不打断的话设计引物就会比较方便。当然如果total RNA本身量比较少,建议老师直接用m6A抗体对total RNA进行富集。
第二步,对RNA进行抗体孵育和特异性富集。打断完了RNA我们会得到长度约为200-300的RNA片段。如果是不打断则是较为完整的RNA全长。这时候我们可以分出约占m6A-IP部分RNA只有10%的RNA作为input。当然如果是细胞样本比较充足,Input和IP的比例可以直接是1:1。如果按照1:1:1,简单来说用于m6A IP的RNA有,3μg,那么Input、IP和IgG的RNA都在1μg左右(无论是total RNA还是mRNA)。
第三步,洗脱和逆转录扩增。接下来我们需要对m6A抗体和IgG抗体上洗脱下来的RNA,以及input的RNA,按照常规试剂盒要求进行洗脱,并用随机引物进行逆转录。
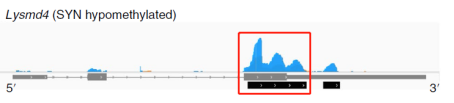
第四步,设计MeRIP-qPCR 的特异性引物。由于m6A甲基化基本富集在UTR区和转录终止区,所以如上图所示在这些区域附近我们可以从测序结果里有明显的peak峰。那么我们设计引物可以考虑在基因高甲基化区域来进行。如果某几个常见的基因在不同文献里频繁出现,那么文献里列出来的引物可以被我们直接拿来引用。通常最后PCR产物长度最低会在130-150bp左右。这里需要注意的是,IP下来的RNA需要用随机引物进行逆转录,而第四步中设计的引物用的是特异性引物,这个大家要注意区分和甄别。
第五步,使用上一步设计好的引物,对input、m6A-IP和IgG-IP中的RNA进行qPCR反应并计算相应的CT值。
所以到这里我们的实验部分已经完成了,简单总结下就是想要做够10个左右基因的验证,必须至少提供>200μg的total RNA以上。富集的RNA类型根据自己要求出发,如polyA RNA或rRNA-depleted RNA等,最后就是打断成200-300nt左右的长度。用于qPCR的RNA一共分成3类,即Input、m6A-IP和IgG-IP,其中input占m6A-IP起始量约10%。如果用数字来表示那就是input需要100ng,m6A在IP前需要1μg,IgG在IP前也需要1μg。如果IP:Input:IgG=1:1:1,则不需要做特别复杂的换算。部分课题组甚至会舍弃IgG,只有IP和Input,分别为1:1。
所以接下来我们就要详细讨论下m6A-IP-qPCR 是如何计算相对表达量的。MeRIP-qPCR 是m6A-IP-qPCR 的另一种说法。所以从名称上我们就能发现,m6A-IP-qPCR基本和常规的RIP-qPCR相似。当然在讨论RIP-qPCR之前,我们先来看看常规的RT-qPCR 是如何计算相对表达量的。
----------------------------------【下载MeRIP-qPCR完整版】--------------------------------
|